查看更多
密码过期或已经不安全,请修改密码
修改密码
壹生身份认证协议书
同意
拒绝

同意
拒绝

同意
不同意并跳过





当地时间9月27日,2019欧洲肿瘤内科学会(ESMO )年会在西班牙巴塞罗那拉开帷幕。今年大会的主题为“将科学研究转化为对癌症患者更好的治疗(translating science into better cancer patient care)”,强调肿瘤领域研究的核心价值在于其转化为治疗手段的潜力。在今年的ESMO大会上,由中国科学院大学附属肿瘤医院(浙江省肿瘤医院)范云教授牵头进行的关于肺癌中枢神经系统转移预测靶点的研究进入了大会口头报告交流,这项研究从DNA甲基化的新视角寻找肺癌CNS转移的预测靶点,引起了全球相关医生及研究者的广泛关注。
 范云教授团队徐艳君主任在ESMO年会现场
范云教授团队徐艳君主任在ESMO年会现场
脑转移是肺癌常见并发症。约10~15%的非小细胞肺癌(NSCLC)患者初诊时伴随脑转移,约30~50%的患者在治疗过程中或病程中会出现脑转移。脑转移的发生率随患者确诊后的生存年限逐年增加;比如EGFR突变患者脑转移发生率在诊疗后1年、2年及3年分别为34.2%, 38.4%和3年46.7%;ALK阳性患者有相似趋势。因此,肺癌脑转移不但是临床医生经常需要面对的一组特殊人群;更让大家感到困惑的是到底什么样的肺癌患者容易出现脑转移。但是,到目前为止,中枢神经系统(CNS)转移发生的机制、遗传背景等尚有待阐明。
研究介绍
入组情况及样本收集
本研究共入组了60例晚期肺癌患者,其中前瞻性收集了18例伴随脑实质转移(BM组)和11例伴随脑膜转移(LM组)的患者,获取患者的肺原发肿瘤组织样本及配对的脑转移样本或脑脊液样本。另回顾性收集了31名随访至因疾病进展死亡均未发生CNS转移(no CNS转移组)的患者,并获取该组患者的肺原发肿瘤组织样本作为对照。
基因检测和甲基化检测
所有样本均使用包含了520个基因的panel(燃石医学,oncoscreen plus)进行基因变异的检测,同时所有样本均使用了燃石医学甲基化测序panel进行甲基化状态的检测。
主要研究结果
基因变异检测:BM组及LM组的原发灶及转移灶样本在驱动基因及TP53的检出上均有高度的一致性。在基因拷贝数变异的检出率上,脑转移样本显著高于原发灶样本。同时,对三组人群肺原发灶样本的基因变异状态进行了比较,结果发现,除了KEAP1基因变异在BM组富集之外,其他基因变异在BM组、LM组及no CNS转移组检出率相当。提示通过基因变异的检测似乎无法找出更容易发生CNS转移的肺癌人群。
甲基化检测:本研究使用的甲基化测序panel包含了超过100000个CpG位点,其中大部分位点位于基因的启动子区域,少部分位点位于基因的外显子、内含子区域,或者基因间序列。同样对三组人群肺原发灶样本的甲基化状态进行了比较;通过对BM组及no CNS转移组的比较,找到了56个存在差异的甲基化区域(blocks);通过对LM组及no CNS转移组的比较,找到了323个存在差异的甲基化区域。将两个比较结果取交集,最终获得了16个blocks。其中15个blocks在BM组及LM组(相比no CNS转移组)具有一致的甲基化变异状态。
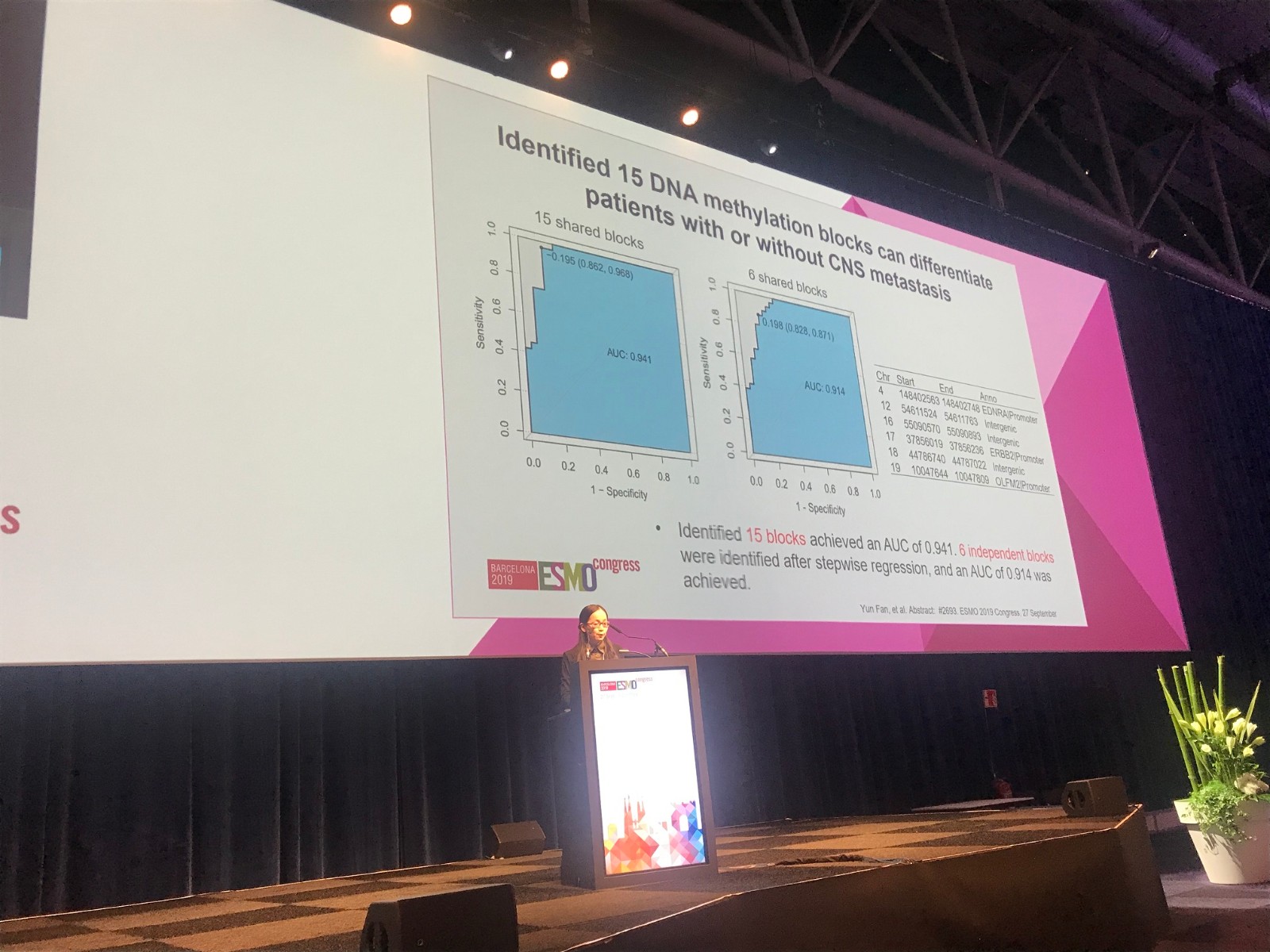
最终通过ROC分析15个blocks以区分发生CNS转移及不发生CNS转移的人群,获得了高达0.941的AUC(area under curve)。通过多因素模型方法,筛选出了6个独立影响的blocks,使用这6个blocks尝试区分BM/LM组及no CNS转移组,获得了高达0.914的AUC。
结论 本研究尝试从基因变异及DNA甲基化两个维度去筛选能够鉴别CNS转移倾向的肺癌人群的预测靶点。结果发现,通过基因变异层面无法实现区分易发生及不发生CNS转移的人群。但是通过甲基化手段,找到了15个DNA甲基化区域能够预测更容易发生CNS转移的肺癌患者。本研究的发现仍需在一个更大的独立人群中进行验证。
作者| 范云 徐艳君 发自西班牙巴塞罗那(中国科学院大学附属肿瘤医院)
编辑 | 郝冉(中国医学论坛报)
查看更多